07 - Ансамблі 🌲🌲🌲🎄🌲
Machine Learning
КНЕУ::ІІТЕ
2023-05-16
Ансамблеві методи
Основи
Замість того, щоб зосереджуватися на навчанні високоточної однієї моделі, ансамблеві методи поєднують багато моделей низької точності в мета-модель.
Три поширені методи для комбінування окремих дерев
- Bagging
- Random forests
- Boosting
Чому? Хоча окремі дерева можуть бути дуже різними та неточними, Комбінація дерев часто досить стабільна і точна.
Bagging
Bagging
Bagging створює додаткові зразки через бутсрап.
Q Як допомагає бутсрап?
A Окремі дерева рішень страждають від варіативності (non-robust).
Ця неробастість означає, що дерева можуть сильно змінюватись в залежності ві того, які спостереження включені/виключені.
По суті, ми використовуємо багато «симуляцій» замість одного набору даних1
Bagging
Bootstrap aggregation (bagging) зменшує цей тип мінливості.
- Створіть \(B\) зразків початкові вибірки з поверненням.
- Навчіть оцінювач (дерево) \(\color{#6A5ACD}{\mathop{\hat{f^b}}(x)}\) на кожній із вибірок \(B\)
- Об’єднайте ваші бустрап моделі \(B\):
\[ \begin{align} \color{#e64173}{\mathop{\hat{f}_{\text{bag}}}(x)} = \dfrac{1}{B}\sum_{b=1}^{B}\color{#6A5ACD}{\mathop{\hat{f^b}}(x)} \end{align} \]
Ця сукупна модель \(\color{#e64173}{\mathop{\hat{f}_{\text{bag}}}(x)}\) є вашою остаточною моделлю.
Bagging
Коли ми застосовуємо баггінг до дерев рішень,
ми зазвичай нарощуємо глибину дерева і не обрізаємо
для регресії ми усереднюємо по регіонах дерев \(B\)
для класифікація ми маємо більше варіантів, але часто беремо більшість
Окремі (необрізані) дерева будуть дуже гнучкими і зашумленими,
але їх узагальнення буде досить стабільним.
Кількість дерев \(B\), як правило, не є критичною для бєггінгу
\(B=100\) в більшості випадків є достатнім.
Out-of-bag error estimation
Бєггінг також пропонує зручний метод оцінки ефективності.
Для будь-якої початкової вибірки ми пропускаємо ~n/3 спостережень.
Out-of-bag (OOB) error estimation оцінює частоту помилок тесту, використовуючи спостереження випадково пропущених з кожного початкового зразка.
Для кожного спостереження \(i\):
- Знайти всі зразки \(S_i\), в яких \(i\) було пропущено у навчальній вибірці
- Узагальнюєте прогнози \(|S_i|\) \(\color{#6A5ACD}{\mathop{\hat{f^b}}(x_i)}\), наприклад, використовуючи їхнє середнє або моду
- Обчисліть похибку, наприклад, \(y_i - \mathop{\hat{f}_{i,\text{OOB},i}}(x_i)\)
Out-of-bag error estimation
Коли \(B\) достатньо великий, частота помилок OOB буде дуже близькою до LOOCV (Leave-One-Out Cross-Validation).
Q Навіщо використовувати коефіцієнт помилок OOB?
A Коли \(B\) і \(n\) великі, перехресна перевірка — з будь-якою кількістю згорток — може стати досить затратною для обчислень.
Ось інструмент для пошуку моделей parsnip:
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Функція: bag_tree()
- “Визначає” модель для
parsnip.
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Функція: bag_tree()
- “Визначає” модель для
parsnip. mode: ‘class.’, ‘reg.’, або unknowncost_complexity: штраф за складність моделі (Cp)tree_depth: макс. глибина дерева
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Функція: bag_tree()
- “Визначає” модель для
parsnip. mode: ‘class.’, ‘reg.’, або unknowncost_complexity: штраф за складність моделі (Cp)tree_depth: макс. глибина дереваmin_n: мін. к-ть спостереж. для поділу
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Функція: bag_tree()
- “Визначає” модель для
parsnip. mode: ‘class.’, ‘reg.’, або unknowncost_complexity: штраф за складність моделі (Cp)tree_depth: макс. глибина дереваmin_n: мін. к-ть спостереж. для поділуclass_cost: збільшення вартості мінорного класу
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Функція: bag_tree()
- “Визначає” модель для
parsnip. mode: ‘class.’, ‘reg.’, або unknowncost_complexity: штраф за складність моделі (Cp)tree_depth: макс. глибина дереваmin_n: мін. к-ть спостереж. для поділуclass_cost: збільшення вартості мінорного класуrpartє движком за замовчуванням
Bagging в R
Ми можемо використовувати tidymodels плюс пакет baguette для бєггінгу дерев.
Функція: bag_tree()
- “Визначає” модель для
parsnip. mode: ‘class.’, ‘reg.’, або unknowncost_complexity: штраф за складність моделі (Cp)tree_depth: макс. глибина дереваmin_n: мін. к-ть спостереж. для поділуclass_cost: збільшення вартості мінорного класуrpartє движком за замовчуваннямtimes: кількість дерев
Приклад: Bagging в R
OOB-based error
# Set the seed
set.seed(12345)
plan(multisession)
# Train the bagged trees
bag_oob = future_map_dfr(
.x = 2:300,
.f = function(n) {
train(
heart_disease ~ .,
data = heart_df,
method = "treebag",
nbagg = n,
keepX = T,
trControl = trainControl(
method = "oob"
)
)$results$Accuracy %>%
data.frame(accuracy = ., n_trees = n)
}
)
# Train the bagged trees
bag_cv <- future_map_dfr(
.x = 2:300,
.f = function(n) {
train(
heart_disease ~ .,
data = heart_df,
method = "treebag",
nbagg = n,
keepX = T,
trControl = trainControl(
method = "cv",
number = 5
)
)$results$Accuracy %>%
data.frame(accuracy = ., n_trees = n)
}
)Bagging та кількість дерев
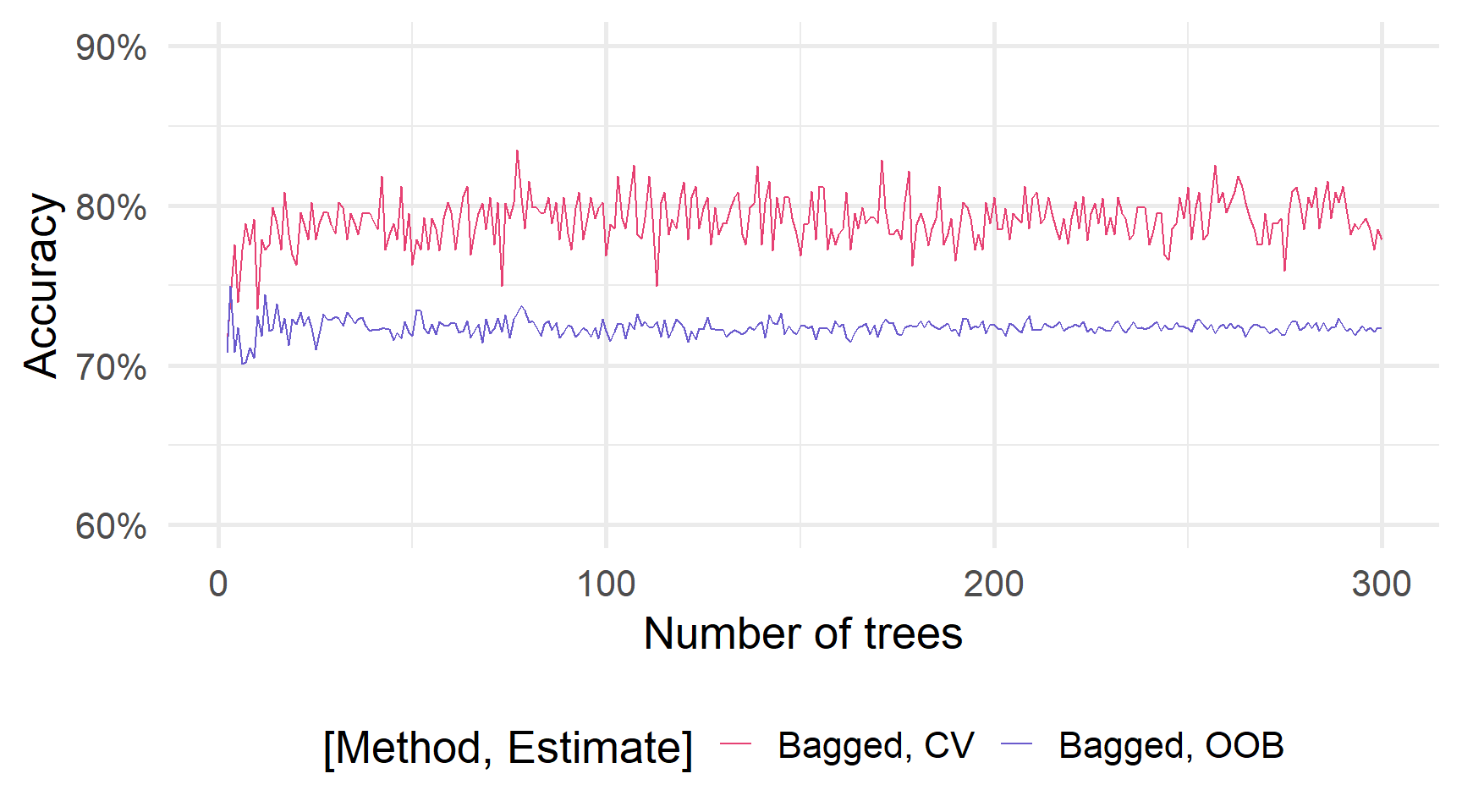
На жаль, ця комбінація rpart/baguette/parsnip/yardstick не пропонує (наразі) показники на основі OOB . 😞
Ми можемо “обдурити” випадкові ліси (ranger) у виконанні OOB для Bagging дерев.
Але спочатку нам потрібно дізнатися про випадкові ліси…
… і перед цим, давайте коротко поговоримо про впливовість змінних.
Впливовість змінних
Впливовість змінних
У той час як ансамблеві методи, як правило, покращують ефективність прогнозування, вони також мають тенденцію знижувати інтерпретованість.
Ми можемо проілюструвати важливість змінних, враховуючи зменшення показників ефективності моделі (RSS, Gini, ентропія тощо)1
У випадку "rpart" bagged дерев…
# Recipe to clean data (impute NAs)
heart_recipe = recipe(heart_disease ~ ., data = heart_df) %>%
step_impute_median(all_predictors() & all_numeric()) %>%
step_impute_mode(all_predictors() & all_nominal())
# Define the bagged tree model
heart_bag = bag_tree(
mode = "classification",
cost_complexity = 0,
tree_depth = NULL,
min_n = 2,
class_cost = NULL
) %>% set_engine(
engine = "rpart",
times = 100
)
# Define workflow
heart_bag_wf = workflow() %>%
add_model(heart_bag) %>%
add_recipe(heart_recipe)
# Fit/assess with CV
heart_bag_fit = heart_bag_wf %>% fit(heart_df)… оцінений об’єкт автоматично включає змінну важливість.
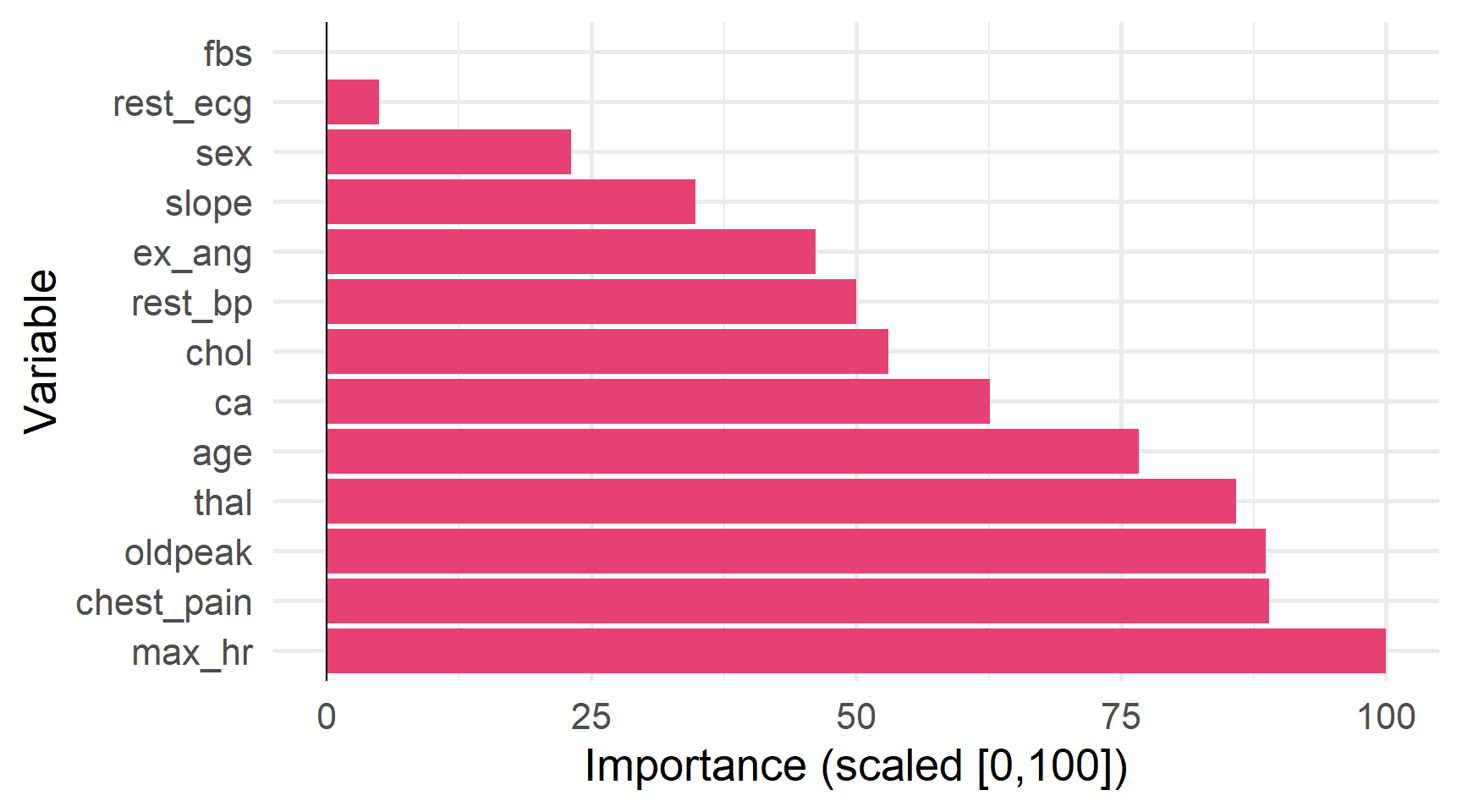
Bagged CART (classification with 100 members)
Variable importance scores include:
# A tibble: 13 × 4
term value std.error used
<chr> <dbl> <dbl> <int>
1 max_hr 41.1 0.839 100
2 chest_pain 36.9 1.18 100
3 oldpeak 36.8 0.806 100
4 thal 35.7 1.35 100
5 age 32.2 0.702 100
6 ca 26.8 1.18 100
7 chol 23.2 0.685 100
8 rest_bp 22.1 0.589 100
9 ex_ang 20.6 0.818 99
10 slope 16.3 0.772 100
11 sex 11.8 0.678 100
12 rest_ecg 4.92 0.311 95
13 fbs 3.02 0.210 93Важливість змінних з нашої bagged tree model.
Bagging
У Bagging є ще один недолік…
Якщо одна змінна домінує над іншими змінними, дерева будуть дуже корельованими.
Якщо дерева дуже корельовані, то bagging втрачає свою перевагу.
Рішення Ми повинні зробити дерева менш корельованими.
Random forests
Random forests
Випадкові ліси покращують bagged trees шляхом декореляції дерев.
Щоб декорелювати дерева, випадковий ліс розглядає лише випадкову підмножину \(\color{#e64173}{m\enspace (\approx\sqrt{p})}\) предикторів при виконанні кожного розбиття (для кожного дерева).
Обмеження змінних, які наше дерево бачить при заданому розділенні:
- не дозволяє деревам постійно використовувати одні і ті ж змінні,
- збільшує різноманітність дерев у нашому лісі,
- потенційно зменшує дисперсію наших оцінок.
Якщо наші прогнози дуже корельовані, ми можемо захотіти зменшити \(m\).
Random forests
Таким чином, випадкові ліси вводять два виміри випадкової варіації
бутсрап вибірка
\(m\) випадково вибрані предиктори (для розбиття)
Все інше про випадкові ліси працює так само, як і з bagging
Random forests в R
У вас є кілька варіантів для навчання випадкових лісів за допомогою tidymodels.
Наприклад, ranger, randomForest, spark.
rand_forest() отримує доступ до кожного з цих пакетів через їх двигуни.
- Механізм за замовчуванням — «ranger» (пакет
ranger).
- Аргумент
mtryдає \(m\), кількість предикторів при кожному розділенні.
Ви вже бачили інші гіперпараметри для ranger:
treesкількість дерев у випадковому лісіmin_nмін. к-ть спостережень
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
Random forests в R
Навчання випадкового лісу в R за допомогою tidymodels…
… і ranger
- Тип: Класифікація
- Три змінні на поділ
- 100 дерев у лісі
- Принаймні 2 спостереж. на поділ
- Движок
ranger - Встановлення правило поділу
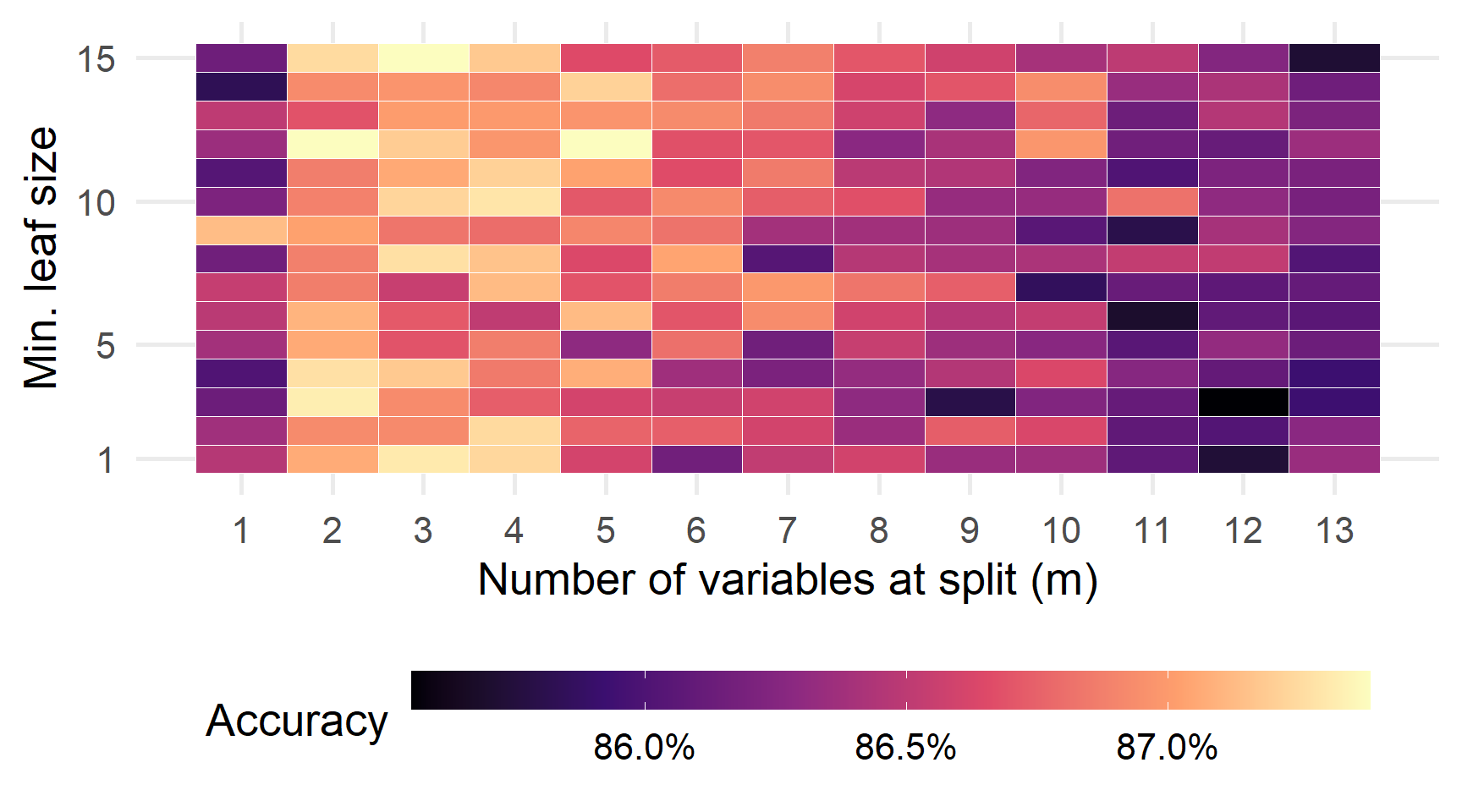
Крок 1: Визначте нашу сітку параметрів
Крок 2: Напишіть функцію, яка оцінює RF, використовуючи задані гіперпараметри.
# Function: One set of hyperparam
rf_i = function(i) {
# Define the random forest
heart_rf_i = rand_forest(
mode = "classification",
mtry = rf_grid[i, 1],
trees = 100,
min_n = rf_grid[i, 2]
) %>% set_engine(engine = "ranger", splitrule = "gini")
# Define workflow
heart_rf_wf_i =
workflow() %>% add_model(heart_rf_i) %>% add_recipe(heart_recipe)
# Fit
heart_rf_fit_i = heart_rf_wf_i %>% fit(heart_df)
# Return DF w/ OOB error and the hyperparameters
return(tibble(
mtry = rf_grid$mtry[i],
min_n = rf_grid$min_n[i],
# Note: OOB error is buried
error_oob = heart_rf_fit_i$fit$fit$fit$prediction.error
))
}Крок 3: Оцініть RF (parallel)!
Точність (OOB) по сітці наших параметрів.
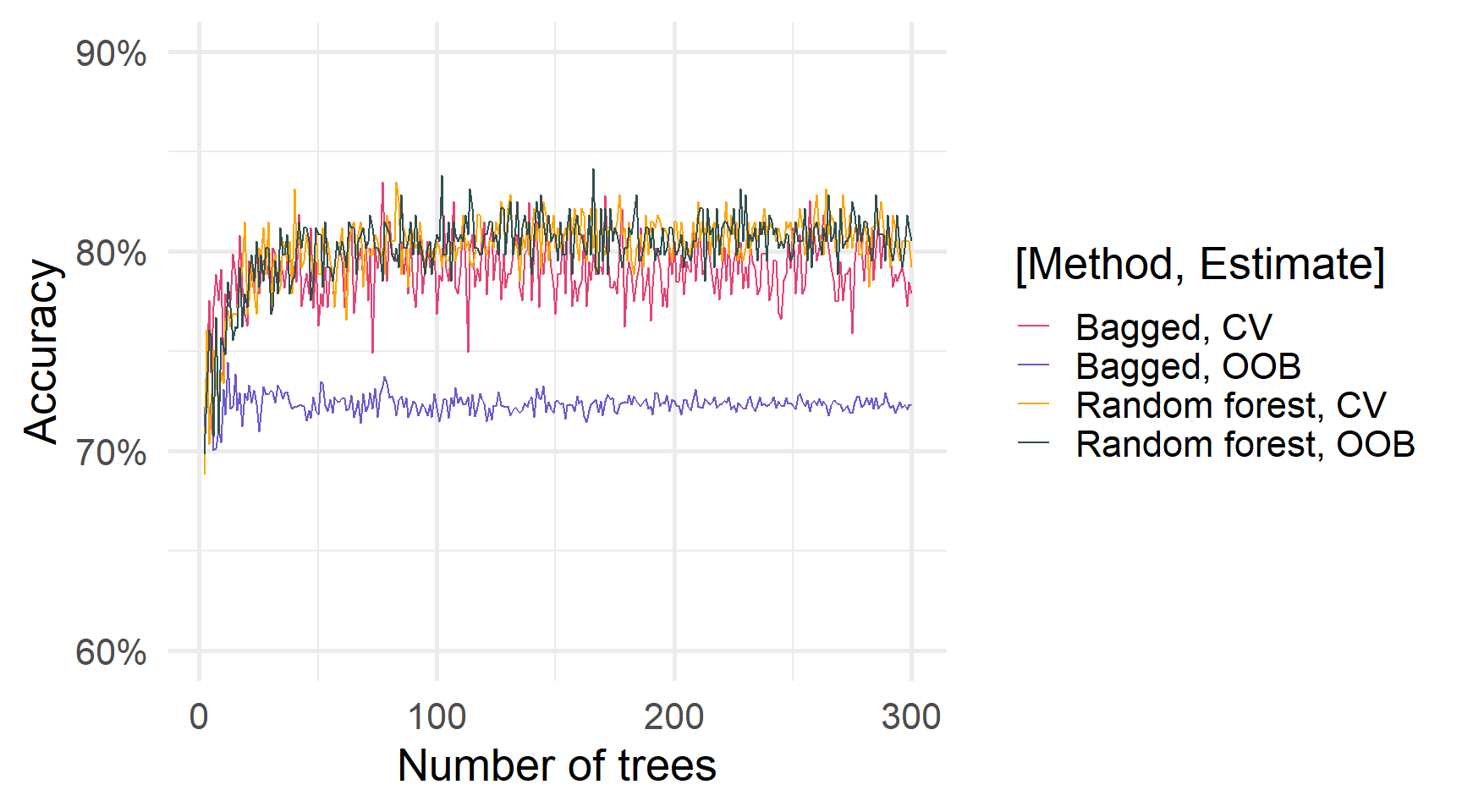
Ансамблі дерев і кількість дерев
Boosting
Boosting
Поки що елементи наших ансамблів виступають самостійно:
жодне окреме дерево нічого не знає про решту лісу.
Boosting дозволяє деревам передавати інформацію одне одному.
Зокрема, boosting навчає свої дерева послідовно — кожне нове дерево тренується на залишках (помилках) своїх попередників.
Ми додаємо кожне нове дерево до нашої моделі \(\hat{f}\) (і оновлюємо наші залишки).
Дерева, як правило, невеликі — повільно покращують \(\hat{f}\).
Boosting
Boosting має три tuning параметри.
- Кількість дерев \(\color{#e64173}{B}\) може бути важливим для запобігання overfitting
2.Параметр зтиснення \(\color{#e64173}{\lambda}\), який контролює швидкість навчання бустінгу (часто 0,01 або 0,001).
- Кількість поділів \(\color{#e64173}{d}\) у кожному дереві (складність дерев).
Окремі дерева, як правило, короткі — часто \(d=1\) («пні»).
Пам’ятайте Дерева вчаться на помилках попередників,
тому жодне дерево не має бути ідеальною моделлю.
How to boost
Крок 1: Установіть \(\color{#6A5ACD}{\mathop{\hat{f}}}(x) = 0\), що дає залишки \(r_i = y_i\) для всіх \(i\).
Крок 2: Для \(\color{#e64173}{b} = 1,\,2\,\ldots,\, B\) виконайте:
A. Підберіть дерево \(\color{#e64173}{\hat{f^b}}\) із розділенням \(d\).
B. Оновити модель \(\color{#6A5ACD}{\hat{f}}\) за допомогою «скороченої версії» нового дерева \(\color{#e64173}{\hat{f^b}}\)
\[ \begin{align} \color{#6A5ACD}{\mathop{\hat{f}}}(x) \leftarrow \color{#6A5ACD}{\mathop{\hat{f}}}(x) + \lambda \mathop{\color{#e64173}{\hat{f^b}}}(x) \end{align} \]
C. Оновіть залишки: \(r_i \leftarrow r_i - \lambda \mathop{\color{#e64173}{\hat{f^b}}}(x)\).
Крок 3: Виведіть розширену модель: \(\mathop{\color{#6A5ACD}{\hat{f}}}(x) = \sum_{b} \lambda \mathop{\color{#e64173}{\hat{f^b}}}(x)\).
Boosted residuals: розширення
Нагадування: Boosting trains - послідовні моделі \(\color{#e64173}{\hat{f_{i}}}(y,x)\) - на залишки попередніх моделей, \(\color{#FFA500}{r_{i-1}}\) (стиснуті на \(\color{#6A5ACD}{\lambda}\))
\[ \begin{align} \color{#FFA500}{r_0} &= y \\[1em] \color{#FFA500}{r_1} &= \color{#FFA500}{r_0} - \color{#6A5ACD}{\lambda} \color{#e64173}{\hat{f_{1}}}(\color{#FFA500}{r_0}, x) \\[0.35em] &= y - \color{#6A5ACD}{\lambda} \color{#e64173}{\hat{f_{1}}}(y, x) \\[1em] \color{#FFA500}{r_2} &= \color{#FFA500}{r_1} - \color{#6A5ACD}{\lambda} \color{#e64173}{\hat{f_{2}}}(\color{#FFA500}{r_1}, x) \\[0.35em] &= y - \color{#6A5ACD}{\lambda} \color{#e64173}{\hat{f_{1}}}(y, x) - \color{#6A5ACD}{\lambda} \color{#e64173}{\hat{f_{2}}}(y - \color{#6A5ACD}{\lambda} \color{#e64173}{\hat{f_{1}}}(y, x), x) \\[1em] &\cdots \end{align} \]
Boosting in R
Ми будемо використовувати boost_tree() з parsnips для тренування boosting дерев1.
boost_tree() приймає кілька параметрів, які ви бачили, плюс ще один:
mtryкількість предикторів для кожного розбиттядерева, кількість дерев \((B)\)min_n, мінімум спостережень для розділенняtree_depth, макс. глибина дереваlearn_rate, швидкість навчання \((\lambda)\)
Boosting in R
# Set the seed
set.seed(12345)
# Train the random forest
heart_boost = train(
heart_disease ~ .,
data = heart_df,
method = "gbm",
trControl = trainControl(
method = "cv",
number = 5
),
tuneGrid = expand.grid(
"n.trees" = seq(25, 200, by = 25),
"interaction.depth" = 1:3,
"shrinkage" = c(0.1, 0.01, 0.001),
"n.minobsinnode" = 5
)
)- boosted за допомогою пакету
gbm
Boosting in R
# Set the seed
set.seed(12345)
# Train the random forest
heart_boost = train(
heart_disease ~ .,
data = heart_df,
method = "gbm",
trControl = trainControl(
method = "cv",
number = 5
),
tuneGrid = expand.grid(
"n.trees" = seq(25, 200, by = 25),
"interaction.depth" = 1:3,
"shrinkage" = c(0.1, 0.01, 0.001),
"n.minobsinnode" = 5
)
)- boosted за допомогою пакету
gbm - перехресна перевірка (без OOB)
Boosting in R
# Set the seed
set.seed(12345)
# Train the random forest
heart_boost = train(
heart_disease ~ .,
data = heart_df,
method = "gbm",
trControl = trainControl(
method = "cv",
number = 5
),
tuneGrid = expand.grid(
"n.trees" = seq(25, 200, by = 25),
"interaction.depth" = 1:3,
"shrinkage" = c(0.1, 0.01, 0.001),
"n.minobsinnode" = 5
)
)- boosted за допомогою пакету
gbm - перехресна перевірка (без OOB)
- CV-пошук пошук по сітці
- кількість дерев
Boosting in R
# Set the seed
set.seed(12345)
# Train the random forest
heart_boost = train(
heart_disease ~ .,
data = heart_df,
method = "gbm",
trControl = trainControl(
method = "cv",
number = 5
),
tuneGrid = expand.grid(
"n.trees" = seq(25, 200, by = 25),
"interaction.depth" = 1:3,
"shrinkage" = c(0.1, 0.01, 0.001),
"n.minobsinnode" = 5
)
)- boosted за допомогою пакету
gbm - перехресна перевірка (без OOB)
- CV-пошук пошук по сітці
- кількість дерев
- глибина дерева
Boosting in R
# Set the seed
set.seed(12345)
# Train the random forest
heart_boost = train(
heart_disease ~ .,
data = heart_df,
method = "gbm",
trControl = trainControl(
method = "cv",
number = 5
),
tuneGrid = expand.grid(
"n.trees" = seq(25, 200, by = 25),
"interaction.depth" = 1:3,
"shrinkage" = c(0.1, 0.01, 0.001), #<<
"n.minobsinnode" = 5
)
)- boosted за допомогою пакету
gbm - перехресна перевірка (без OOB)
- CV-пошук пошук по сітці
- кількість дерев
- глибина дерева
- швидкість навчання
Boosting in R
# Set the seed
set.seed(12345)
# Train the random forest
heart_boost = train(
heart_disease ~ .,
data = heart_df,
method = "gbm",
trControl = trainControl(
method = "cv",
number = 5
),
tuneGrid = expand.grid(
"n.trees" = seq(25, 200, by = 25),
"interaction.depth" = 1:3,
"shrinkage" = c(0.1, 0.01, 0.001),
"n.minobsinnode" = 5 #<<
)
)- boosted за допомогою пакету
gbm - перехресна перевірка (без OOB)
- CV-пошук пошук по сітці
- кількість дерев
- глибина дерева
- швидкість навчання
- мінімальний розмір листка
Порівняння boosting параметрів — зверніть увагу на швидкість навчання
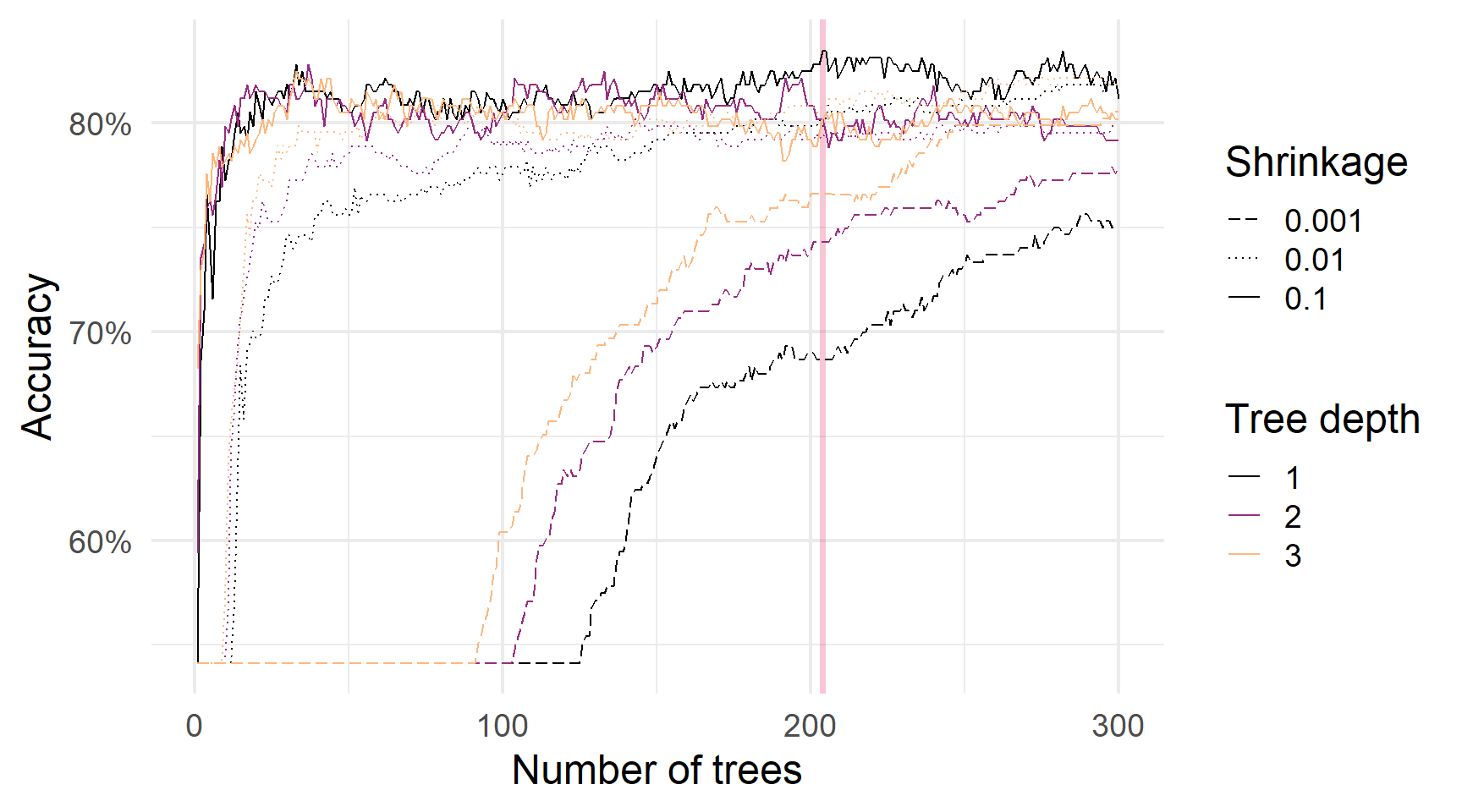
Порівняння boosting параметрів — більше дерев
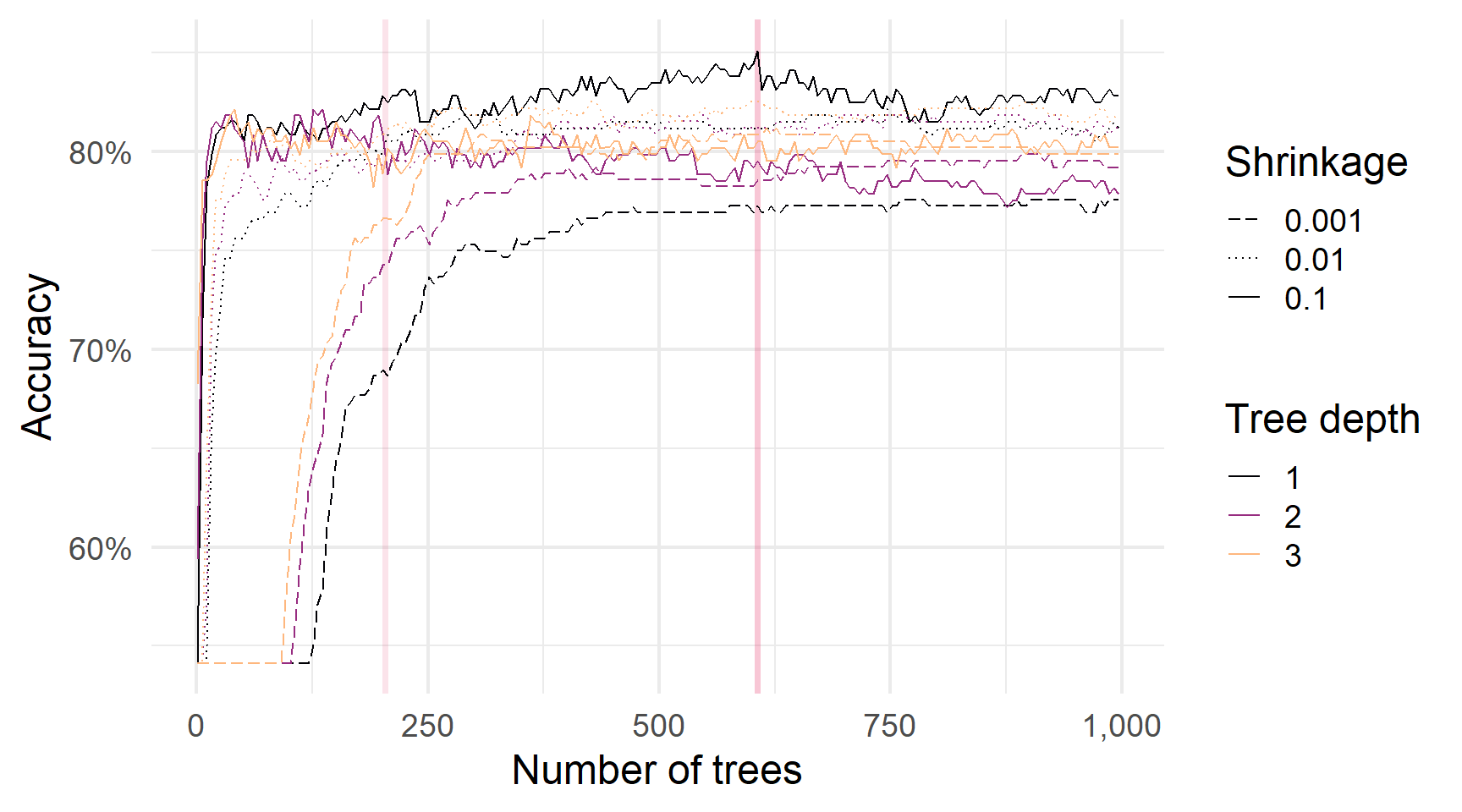
Порівняння boosting параметрів — ще більше дерев
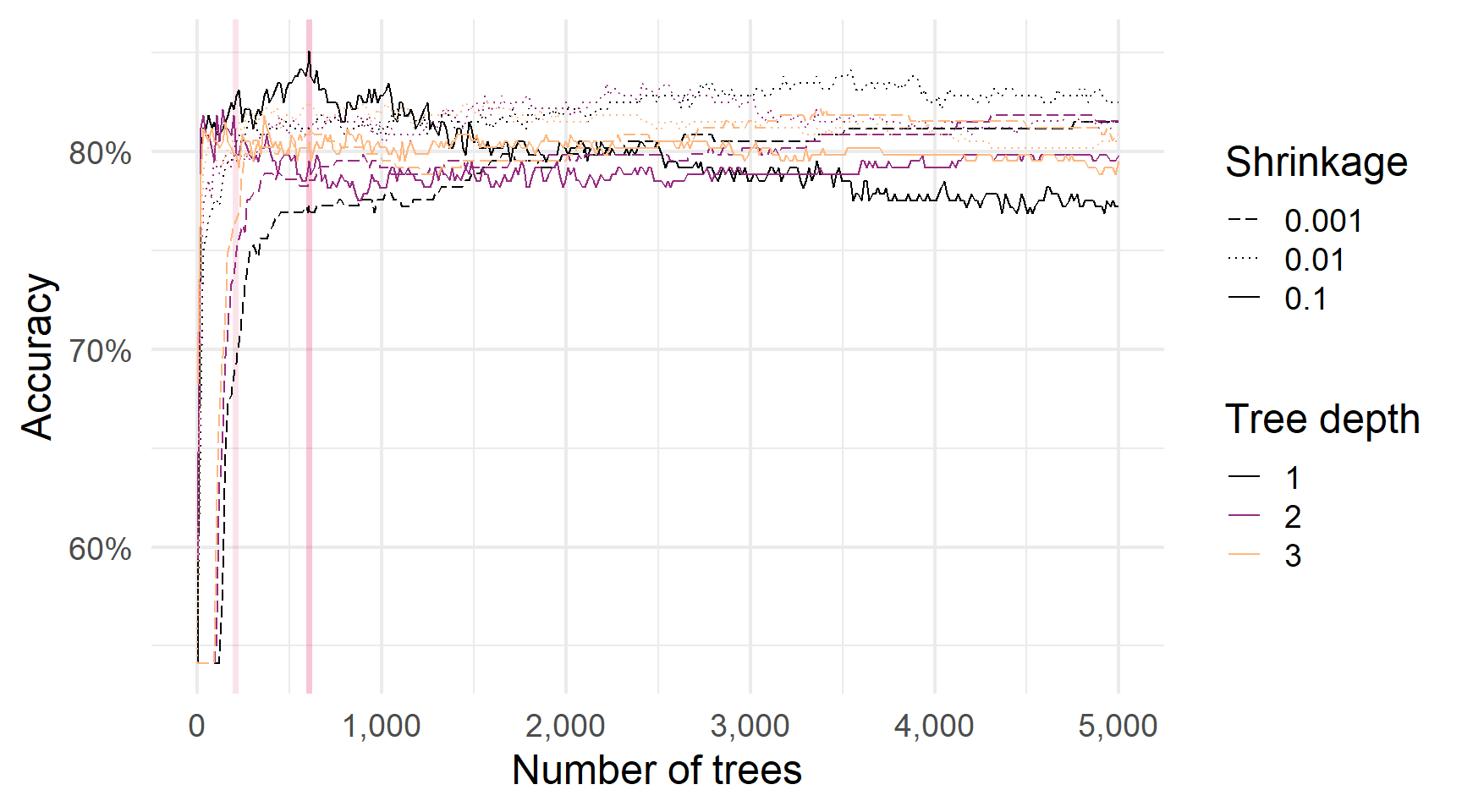
Порівняння boosting параметрів — і ще більше дерев
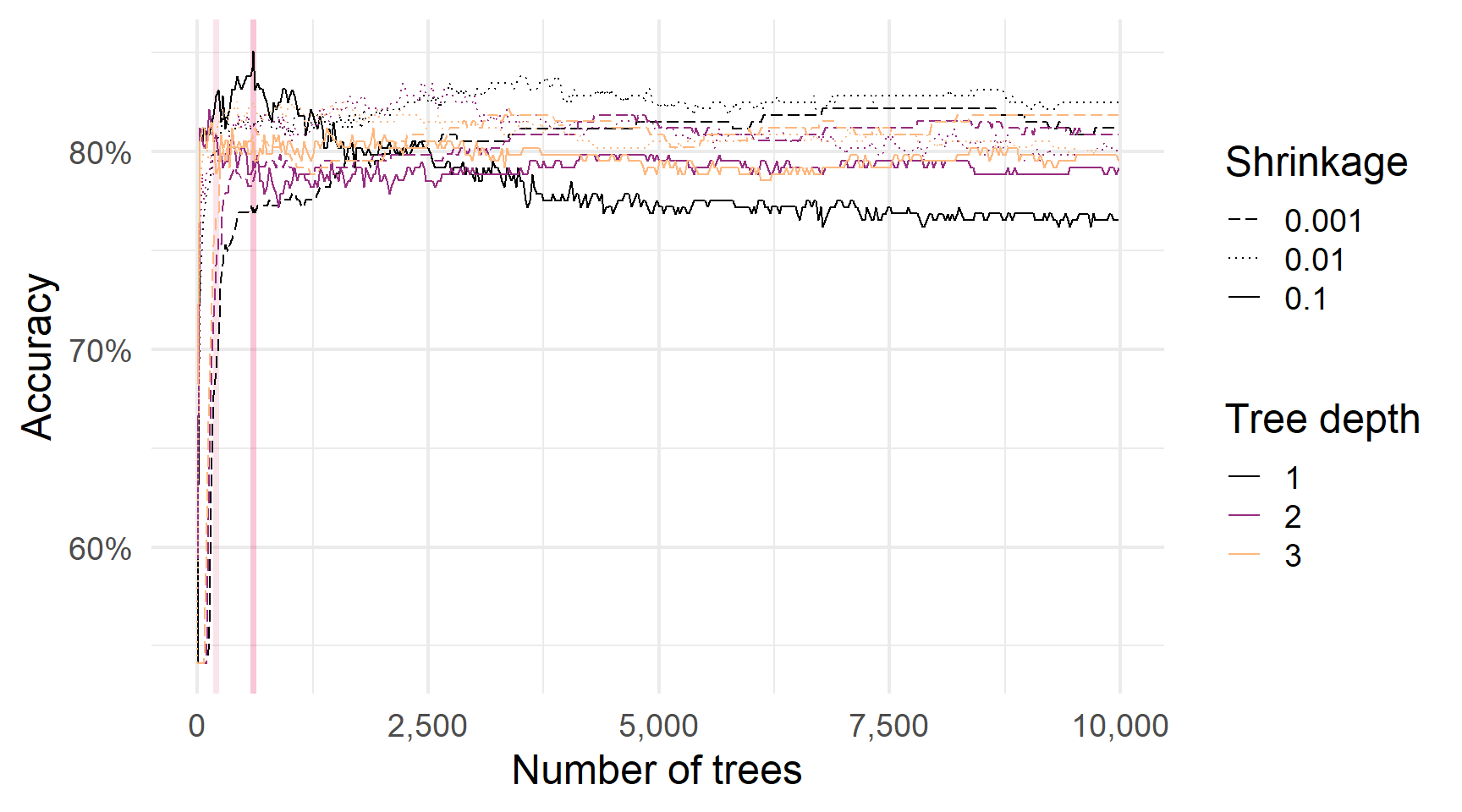
Ансамблі дерев та кількість дерев
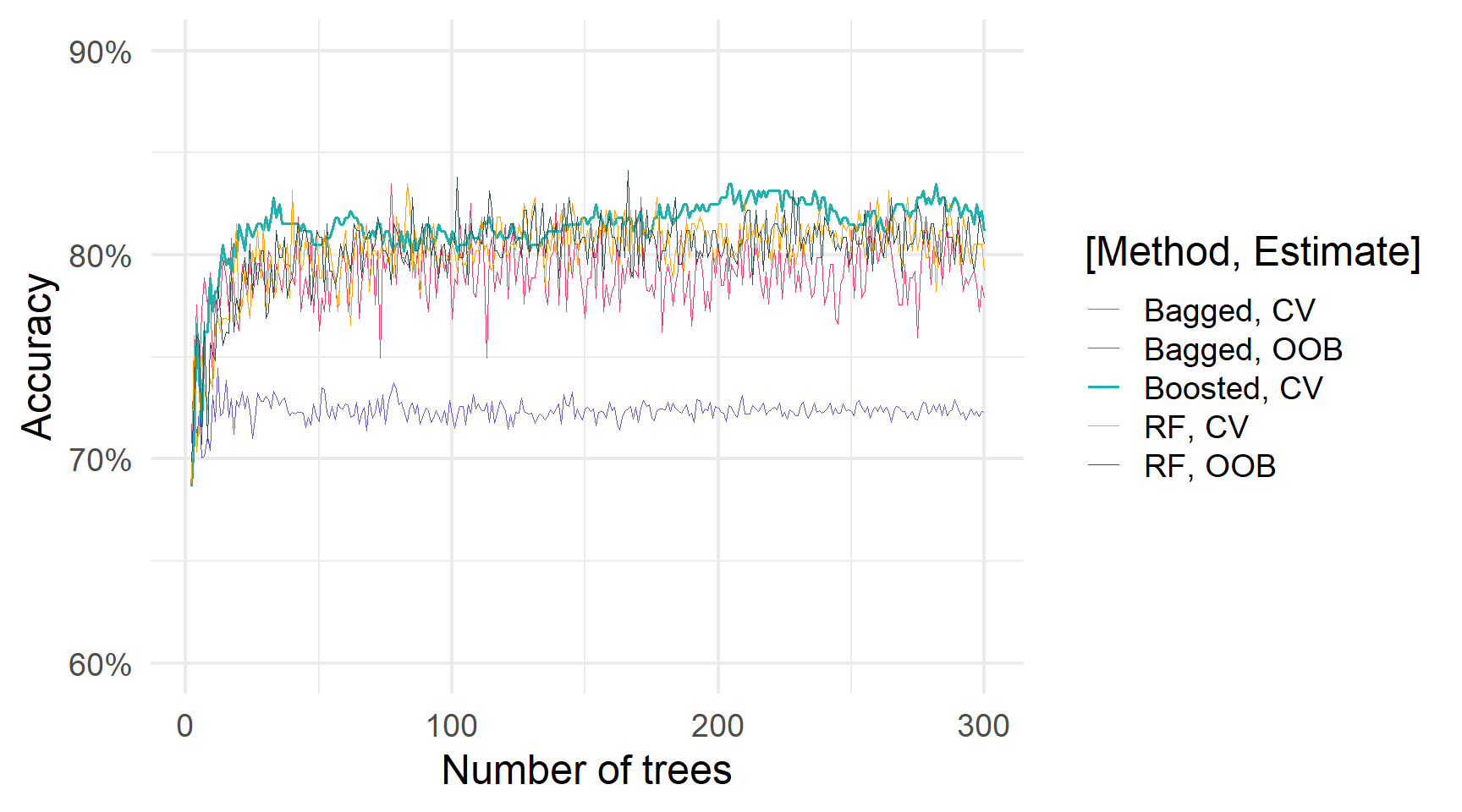
Звичайно, є багато інших варіантів навчання на основі дерев:
Дякую за увагу!
ihor.miroshnychenko@kneu.ua
